- ⌂
-
Le DPM
Les Equipements
L'Environnement
Zoom sur... Le Bâtiment André Rassat
Nommé d'après une figure tutélaire de la chimie grenobloise, ce bâtiment est recouvert d’une double peau en feuille métallique qui apporte une protection thermique sur 3 côtés et crée une unité architecturale favorisant l'intégration parmi les arbres du site.
-
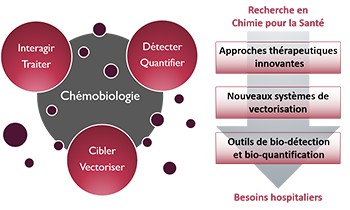
Thématiques
Le Thème
« Approches thérapeutiques innovantes »De nouvelles solutions thérapeutiques, de la cible biomacromolécu-laire émergente aux approches originales pour traiter les maladies
Le Thème
« Nouveaux systèmes de vectorisation »Combiner les propriétés d’inclusion de principes actifs, de franchisse-ment de barrières, d’adressage et de relar-gage en milieu vivant
Le Thème
« Outils de bio-détection et bio-quantification »Des dispositifs analytiques originaux pour la détection de cibles, de l’ion au micro-organisme en milieu complexe
Zoom sur... La Chémobiologie
-
Équipes
L'Équipe « COMET »
« COMET » développe la conception rationnelle, la synthèse et/ou l'extraction de composés à forte diversité/complexité comme nouveaux agents thérapeutiques et outils moléculaires pour la pénétration cellulaire ou la détection de biomolécules, actifs in vivo.L'Équipe « NOVA »
« NOVA » utilise des acides nucléiques fonctionnels comme éléments de reconnaissance pour des applications thérapeutiques ou diagnostiques, comme la sélection d'oligonucléotides, ou le développement de dispositifs d'analyses et de nanovecteurs.Les Services
-
Productions
Les Publications
La Vulgarisation
Les JSM
Zoom sur... La 12ème JSM (15 juin 2023)
Le DPM organise des journées scientifiques consacrées au médicament. L'objectif est de rassembler les spécialistes académiques et industriels autour d'une thématique. 2023 : Apports de la Chimie Click et de la Lumière en Chemobiologie
-
Partenariats
Les Formations
Les Consortiums
Les Financements
Zoom sur... L'environnement Grenoblois
Le DPM est un acteur central sur le bassin grenoblois en chimie, biologie et santé, lié au CHU Grenoble Alpes et à de nombreuses autres organisations : Pole de Recherche CBS, ICMG, Labex ARCANE, EUR CBH, Institut Carnot Polynat, Réseau GREEN.
Drug design rationnel et cibles thérapeutiques complexes
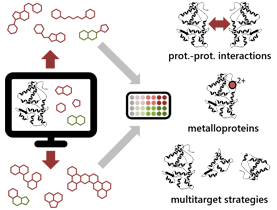
Le DPM dispose d'expertises et d'infrastructures dédiées au support des activités de « drug design » rationnel construit à partir d'une cible identifiée. L'emphase est placée dans ce projet sur l'optimisation et la pharmacomodulation de composés pour des approches émergentes impliquant des cibles et des modes d'interaction complexes, comme les métalloenzymes, les interactions protéine-protéine ou la mise en place de stratégies multi-cibles. Dans ce cadre, l'apport de la modélisation moléculaire (docking et dynamique moléculaires) est particulièrement précieux ; la participation d'un membre de l'équipe spécialisé dans les approches rationnelles et chémoinformatiques permet d'assurer la robustesse et la pertinence des travaux menés.
Les métalloenzymes, notamment, constituent des cibles souvent difficiles à traiter en modélisation moléculaire, à cause de la présence de métaux de transitions aux propriétés électroniques mal décrites par les logiciels classiques. Plusieurs stratégies sont donc élaborées dans le cadre du projet afin de surmonter ces difficultés : la combinaison d'un criblage virtuel de fragments en QM/MM avec un criblage réel réalisé sur la protéine et observé par RMN (ciblage de NDM-1, résistance bactérienne), ou la modélisation en QM/MM des interactions entre potentiels inhibiteurs et modèles d'homologie (ciblage de la tyrosinase humaine, mélanome et applications dermocosmétiques), par exemple (axe « Combinaison de stratégies pour cibler les métalloenzymes »).
Les spécificités des approches basées sur la structure pour identifier des inhibiteurs d'interactions protéine-protéine sont également peu supportées par les logiciels classiques de modélisation. Plusieurs stratégies sont donc explorées. Une première approche combinant criblage haut débit et RMN permet l'identification d'inhibiteurs de l'interface TFPI–FXa. Une seconde étude s'intéresse à la sélectivité d'inhibiteurs de BET (BD2 vs. BD1) à travers l'étude de ligands co-cristallisés et des données issues de la littérature (axe « Découverte d'inhibiteurs sélectifs des interactions protéine-protéine »).
Enfin, les ligands conçus pour atteindre plusieurs cibles à la fois offrent un nouvel espoir de traitement pour des pathologies multifactorielles complexes comme la maladie d'Alzheimer (AD). Les cibles dont la structure a été résolue sont en particulier considérées dans ce projet, comme les cholinestérases ou les peptides amyloïdes. L'optimisation d'une série d'hybrides indolizine-pyridium, possédant déjà plusieurs activités dans le cadre d'AD, a pour objectif d'inclure de nouvelles cibles dans l'approche, comme BACE-1, MAO ou GSK-3, avec l'aide du docking moléculaire (axe « Exploration de stratégies multi-cibles pour le traitement de pathologies complexes »).
Jean-Luc DECOUT, Yung-Sing WONG, Martine DEMEUNYNCK, Isabelle BAUSSANNE, Marine PEUCHMAUR, Basile PERES, Benjamin BOUCHERLE, Romain HAUDECOEUR, Aline THOMAS, Matis MORETTI
Mots-clés : Dynamique Moléculaire, In silico Drug Design, Pharmacomodulation, Modélisation Moléculaire, Cancer, Antibiotiques, Chimiorésistance, Mucoviscidose, Hétérocycles, Cibles difficiles
Publications
-
Article • Comparative Metabolomics Reveals How the Severity of Predation by the Invasive Insect Cydalima Perspectalis Modulates the Metabolism Re–Orchestration of Native Buxus Sempervirens [Full paper
 ]
] Hay, A. E.; Deborde, C.; Dussarrat, T.; Moing, A.; Millery, A.; Hoang, T. P. T.; Touboul, D.; Rey, M.; Ledru, L.; Ibanez, S.; Pétriacq, P.; Vanhaverbeke, C.; Gallet, C.
Plant Biology 2025, 27 (5), 818–833
-
Article • ABCB4 Disease-Causing Variants S242R, S346I, T437I and T1077M Significantly Impair Its Function and Display Differential Sensitivity to Potentiators [Full paper
 ]
] Madry, C.; Elbahnsi, A.; Delaunay, J.-L.; Stary, A.; Lagaye, S.; Couvert, P.; Corpechot, C.; Lemoinne, S.; Chignard, N.; Boucherle, B.; Gautheron, J.; Décout, J.-L.; Callebaut, I.; Aït-Slimane, T.
Sci Rep 2025, 15 (1), 44544
-
Article • A Dual-Affinity Peptide Nucleic Acid for Targeting miRNA-21 Precursor Rescues Tumor Repressor PTEN Expression [Full paper
 ]
] Lian, Y.; Wang, A.; Lin, R.; Ke, X.; Zhan, X.; Lu, R.; Qian, T.; Ghosh, A.; Ong, A. A. L.; Toh, D.-F. K.; Patil, K. M.; Krishna, M. S.; Dezanet, C.; Decout, J.-L.; Maiti, S.; Wang, H.; Chen, G.
Cell Reports Physical Science 2025, 6 (11), 102935
-
Article • The Thienopyrimidinone Gamhépathiopine Targets the QO Site of Plasmodium Falciparum Cytochrome b [Full paper
 ]
] Wiedemar, N.; Milne, R.; Carvalho, S.; Patterson, S.; Bodkin, M.; Masurier, N.; Lisowski, V.; Primas, N.; Verhaeghe, P.; Sloan, G. M.; Wyllie, S.
ACS Infect. Dis. 2025, 11 (6), 1719–1728
-
Article • Aurones as New Modulators of Hypoxia Inducible Factor-1 (HIF-1) to Prevent Cardiac Cell Death [Full paper
 ]
] Di Maria, L.; Roulier, B.; Beaumet, M.; Haudecoeur, R.; Boumendjel, A.; Belaïdi, E.; Pérès, B.
Results Chem. 2025, 17, 102609
-
Article • Hydroquinone Causes Exogenous Ochronosis through Tyrosinase Catalytic Activity and Highlights the Need for Genuine Human Tyrosinase Inhibitors [Full paper
 ]
] Beaumet, M.; Haudecoeur, R.
Br. J. Dermatol 2025, 193 (5), 817–818
-
Article • Effect of Bromodomain and Extraterminal Inhibitors with Different Bromodomain Selectivity on Mesenchymal Stem Cells [Full paper
 ]
] Tanimoto, R.; Wong, Y.-S.; Ebara, M.; Uto, K.
Chem. Lett. 2025, 54 (1), upae238
-
Article • Design and synthesis of 4-amino-2′,4′-dihydroxyindanone derivatives as potent inhibitors of tyrosinase and melanin biosynthesis in human melanoma cells [Full paper
 ]
] L. M. Lazinski, M. Beaumet, B. Roulier, R. Gay, G. Royal, M. Maresca, R. Haudecoeur
European Journal of Medicinal Chemistry, 2024, 266, pp.116165. ⟨10.1016/j.ejmech.2024.116165⟩
-
Article • Aurones and derivatives as promising New Delhi metallo-β-lactamase (NDM-1) inhibitors [Full paper
 ]
] Jérémy Caburet, Federica Verdirosa, Matis Moretti, Brayan Roulier, Giorgia Simoncelli, Romain Haudecoeur, Somayeh Ghazi, Hélène Jamet, Jean-Denis Docquier, Benjamin Boucherle, Marine Peuchmaur
Bioorganic & Medicinal Chemistry 2024 97, 117559
-
Article • Enantioselective Approach for Expanding the Three-Dimensional Space of Tetrahydroquinoline to Develop BET Bromodomain Inhibitors [Full paper
 ]
] Marie-Ange Lespinasse, Kaiyao Wei, Justine Perrin, Matthias Winkler, Sieme Hamaidia, Alexis Leroy, Zuzana Macek Jilkova, Christian Philouze, Patrice N. Marche, Carlo Petosa, Jérôme Govin, Anouk, Emadali, Yung-Sing Wong
Chem. Eur. J. 2022, 28, e202202293
-
Article • Bioactive Aurones, Indanones, and Other Hemiindigoid Scaffolds: Medicinal Chemistry and Photopharmacology Perspectives [Full paper
 ]
] Leticia M. Lazinski, Guy Royal, Maxime Robin, Marc Maresca, Romain Haudecoeur
J. Med. Chem. 2022, 65, 12594 625
-
Article • Exploring the Structure-Activity Relationship of Benzylidene-2,3-Dihydro-1H-Inden-1-One Compared to Benzofuran-3(2H)-One Derivatives as Inhibitors of Tau Amyloid Fibers [Full paper
 ]
] Emeline Boukherrouba, Camille Larosa, Kim-Anh Nguyen, Jérémy Caburet, Laurent Lunven, Hugues Bonnet, Antoine Fortuné, Ahcène Boumendjel, Benjamin Boucherle, Sabine Chierici, Marine Peuchmaur
Eur. J. Med. Chem. 2022, 231, 114139
-
Article • A fragment-based drug discovery strategy applied to the identification of NDM-1 β-lactamase inhibitors [Full paper
 ]
] Jérémy Caburet, Benjamin Boucherle, Sofiane Bourdillon, Giorgia Simoncelli, Federica Verdirosa, Jean-Denis Docquier, Yohann Moreau, Isabelle Krimm, Serge Crouzy, Marine Peuchmaur
European Journal of Medicinal Chemistry 240 (2022) 114599
-
Patent • Method for synthesizing diversely substituted purines
Jean-Luc Decout, Renaud Zelli, Wael Zeinyeh, Benjamin Boucherle, Romain Haudecoeur
2021 •
-
Article • Ditopic Chelators of Dicopper Centers for Enhanced Tyrosinases Inhibition [Full paper
 ]
] E. Buitrago, C. Faure, L. Challali, E. Bergantino, A. Boumendjel, L. Bubacco, M. Carotti, R. Hardré, M. Maresca, C. Philouze, H. Jamet, M. Réglier, C. Belle
Chem. Eur. J. 2021, 27, 43844393
-
Article • Interest of Homodialkyl Neamine Derivatives against Resistant P. Aeruginosa, E. Coli, and β-Lactamases-Producing Bacteria—Effect of Alkyl Chain Length on the Interaction with LPS [Full paper
 ]
] J. Swain, C. Dezanet, H. Chalhoub, M. Auquière, J. Kempf, J.-L. Décout, M.-P. Mingeot-Leclercq
Int. J. Mol. Sci. 2021, 22, 8707
-
Article • β-Carboline as a Privileged Scaffold for Multitarget Strategies in Alzheimer’s Disease Therapy [Full paper
 ]
] A. Beato, A. Gori, B. Boucherle, M. Peuchmaur, R. Haudecoeur
J. Med. Chem. 2021, 64, 13921422
-
Article • Pharmacological Chaperones Improve Intra-Domain Stability and Inter-Domain Assembly via Distinct Binding Sites to Rescue Misfolded CFTR [Full paper
 ]
] N. Baatallah, A. Elbahnsi, J.-P. Mornon, B. Chevalier, I. Pranke, N. Servel, R. Zelli, J.-L. Decout, A. Edelman, I. Sermet-Gaudelus, I. Cellebaut, A. Hinzpeter
Cell. Mol. Life Sci. 2021, 78, 7813-7829
-
Article • Interest of novel N-alkylpyridinium-indolizine hybrids in the field of Alzheimer’s disease: Synthesis, characterization and evaluation of antioxidant activity, cholinesterase inhibition, and amyloid fibrillation interference [Full paper
 ]
] I. Baussanne, O. Firstova, A. Botezatu Dediu, C. Larosa, B. Furdui, I. Ottilia Ghinea, A. Thomas, S. Chierici, R. Dinica, M. Demeunynck
Bioorg. Chem. 2021, 116, 105390
-
Article • Chromones bearing amino acid residues: Easily accessible and potent inhibitors of the breast cancer resistance protein ABCG2 [Full paper
 ]
] E. Roussel, A. Moréno, N. Altounian, C. Philouze, B. Pérès, A. Thomas, O. Renaudet, P. Falson, A. Boumendjel
Eur. J. Med. Chem. 2020, 202, 112503
-
Article • Advances in the Design of Genuine Human Tyrosinase Inhibitors for Targeting Melanogenesis and Related Pigmentations [Full paper
 ]
] Brayan Roulier, Basile Pérès, Romain Haudecoeur
J. Med. Chem. 2020, 63, 22, 13428–13443
-
Article • Targeting different binding sites in the CFTR structures allows to synergistically potentiate channel activity [Full paper
 ]
] L. Froux, A. Elbahnsi, B. Boucherle, A. Billet, N. Baatallah, B. Hoffmann, J. Alliot, R. Zelli, W. Zeinyeh, R. Haudecoeur, A. Fortuné, S. Mirval, C. Simard, P. Lehn, J.-P. Mornon, I. Callebaut, J.-L. Décout
Eur. J. Med. Chem. 2020, 190, 112116
-
Patent • Compounds for treating cystic fibrosis
Isabelle Callebaut, Jean-paul Mornon, Jean-Luc Decout, Frederic Becq, Pierre Lehn, Brice Hoffman, Benjamin Boucherle, Romain Haudecoeur, Antoine Fortune, Clement Boinot, Julien Alliot
2019 •




 Annuaire
Annuaire Contact
Contact Plan d'accès
Plan d'accès ENG
ENG Login
Login




